Già da novembre 2019 erano state rilevati decine di casi di polmoniti atipiche a Wuhan, la città più popolosa della Cina orientale, centro commerciale estremamente attivo ed il 31 dicembre ne era stata data la notizia ufficiale. Dalle prime indagini era emerso che i contagiati erano tutti frequentatori assidui del mercato Huanan Seafood Wholesale Market a Wuhan, che pertanto era stato chiuso dal 1 gennaio 2020.
Questo sito usa i servizi di Analytics e Marketing Automation ShinyStat™
Privacy policy per i visitatori dei siti che usano i “servizi di ShinyStat”
Si stava già facendo strada l’ipotesi che la “causa” avesse un’origine animale (zoonosi) connessa con il mercato.
Il 7 gennaio il virus responsabile veniva isolato e pochi giorni dopo, il 12 gennaio, il suo genoma era sequenziato e il risultato condiviso con il resto del mondo, dando la possibilità di approntare kit diagnostici.
Il 9 gennaio le autorità cinesi avevano annunciato che il patogeno responsabile era un nuovo ceppo di coronavirus, della stessa famiglia dei coronavirus responsabili della SARS del 2003 e della MERS del 2012, ma diverso da questi. La notizia era ripresa dall’Oms il 10 gennaio, fornendo istruzioni di massima per minimizzare il contagio.
Il 21 gennaio le autorità sanitarie locali cinesi e l’OMS annunciavano che il nuovo coronavirus, passato probabilmente dall’animale (pipistrello?) all’essere umano (un salto di specie, spillover), si poteva trasmettere anche da uomo a uomo. L’intera regione di Wuhan veniva isolata per bloccare il contagio, mentre nel resto del mondo veniva solo raccomandato di non recarsi in Cina (non viceversa) salvo situazioni di necessità.
Infatti, in Italia i primi casi del 31 gennaio erano soggetti provenienti dalla Cina, due turisti cinesi di Wuhan, e un ricercatore italiano positivo al virus e proveniente dalla Cina.
Solo alla fine di gennaio l’OMS dichiarava l’emergenza sanitaria pubblica di interesse internazionale e l’Italia bloccava i voli da e per la Cina, unica in Europa, quando la situazione cinese stava già migliorando. Il nuovo virus verrà battezzato SARS-CoV-2, mentre la malattia CoViD-19.
La partita Atalanta-Valencia al San Siro del 19 febbraio ha di fatto costituito l’occasione di un contagio diffuso Italia-Spagna dato l’inizio all’impennata dei mesi successivi.
Il 21 febbraio 2020 sono emersi diversi casi di coronavirus nel lodigiano, in Lombardia e poi nel Veneto. Alcuni dei paesi colpiti (Codogno, Castiglione d’Adda e Casalpusterlengo ed altri) sono stati di fatto chiusi dal governo, creando le rimostranze delle forze politiche d’opposizione. Per alcune settimane gli scienziati e gli opionionisti si sono divisi fra apocalittici e minimizzatori, dando vita ad un’informazione contraddittoria che ha rallentato l’applicazione delle misure di isolamento e quindi le possibilità di contenere l’epidemia.
Anche nel resto dell’Europa e negli altri Paesi i tempi di reazione all’epidemia, poi elevata a pandemia dal 11/03/2020, sono stati molti lenti, portando alla situazione attuale.
In Italia mercoledì 4 marzo il governo ha dato il via libera alla chiusura di scuole e università in tutta Italia fino al 15 marzo, domenica 8 marzo ha ordinato l’isolamento della Lombardia e di altre 14 province, che diventano “zona rossa”, lunedì 9 marzo è stato esteso a tutto il paese le misure già prese per la Lombardia e per le altre 14 province. Di fatto la regola è contenuta nell’hashtag #iorestoacasa, si può uscire solo per comprovate ragioni di necessità come per fare la spesa, per esigenze lavorative, per l’acquisto di farmaci o per altri motivi di salute. Con domenica 22 marzo, sono state chiuse tutte le attività non essenziali alla vita delle persone.
Per seguire l’andamento della pandemia in Italia si può far riferimento al sito del Ministero della salute: http://www.salute.gov.it/portale/news/p3_2_1_1_1.jsp?lingua=italiano&menu=notizie&p=dalministero&id=4297
I coronavirus
Sono virus la cui informazione genetica risiede in un filamento di RNA, particolarmente grande. L’acido ribonucleico è contenuto in un capside, una struttura costituita dalla ripetizione di subunità proteiche, dette capsomeri (i mattoni), a loro volta formate dalla ripetizione di proteine virali.
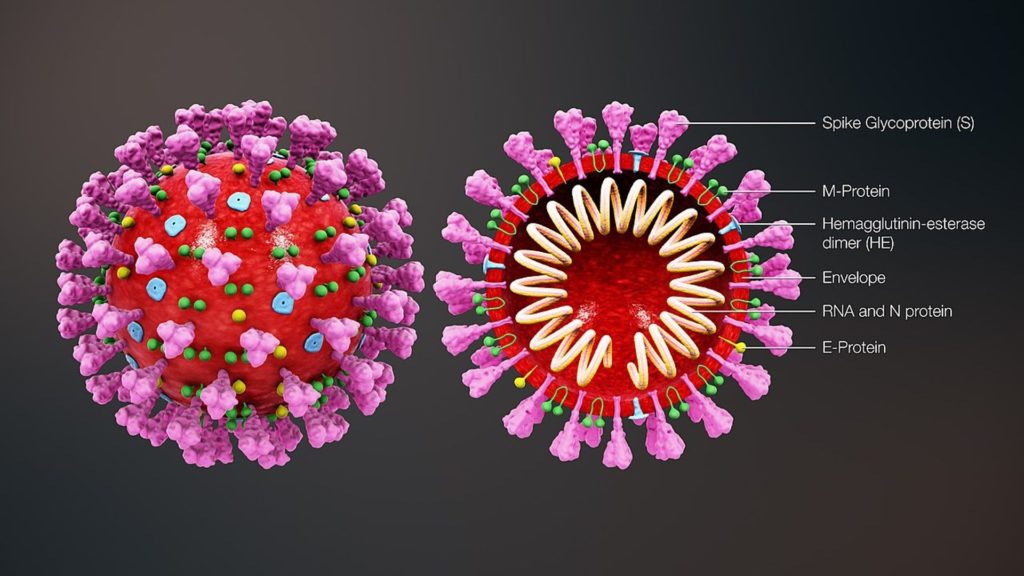
Il capsomero è rivestito da una membrana più esterna, chiamata pericapside (peplos, mantello, envelope), che deriva dalla membrana plasmatica della cellula ospite e come tale è costituita principalmente da fosfolipidi, polisaccaridi e glicoproteine. I virus dotati di pericapside sono chiamati “virus rivestiti” e generalmente possiedono anche una struttura di natura proteica, detta matrice o tegumento, localizzata tra il capside ed il pericapside.
Al microscopio elettronico appaiono come un corpo rotondeggiante da cui emanano estroflessioni, peplomeri (proteina S), ad estremità allargata come la testa di un chiodo, che danno al virus l’aspetto di una corona, da cui il nome.
La proteina S è la chiave che, interagendo con l’aminopeptidasi N della membrana mediante uno specifico recettore a cui si lega, apre l’accesso alle cellule che può infettare. La proteina S stimola la produzione di anticorpi contro il virus, che per il resto è protetto dal pericapside.
Per vedere lo schema strutturale si può far riferimento alla seguente figura tratta da: https://www.scientificanimations.com/wiki-images/.
I coronavirus erano già stati identificati come i responsabili di riniti comuni oltre 50 anni fa. Viceversa i due ceppi identificati nel 2003 e nel 2012 determinarono gravi infezioni delle basse vie respiratorie per cui furono denominati SARS-CoV (Sindrome acuta respiratoria severa) e MERS-CoV (Sindrome respiratoria medio-orientale). La prima proveniente probabilmente da pipistrelli, ha determinato circa 8.000 casi nel mondo con 800 deceduti, la seconda proveniente dai dromedari ha infettato circa 1500 persone in Arabia Saudita con 500 morti.
Tutti i virus hanno un nucleo formato da materiale genetico, cioè da un acido nucleico, che può essere DNA oppure RNA e sono pertanto denominati desossiribovirus e ribovirus. Il genoma può essere costituito da 2.000-200.000 basi nucleotidiche per l’espressione di 5-100 geni (proteine/enzimi).
Il DNA può essere:
- a doppia catena (dsDNA)
- a doppia catena circolare
- a singola catena (ssDNA).
L’ RNA, a sua volta, si trova:
- a singola catena con intermedio a DNA,
- a singola catena intera, positiva o negativa, (+ssRNA, -ssRNA)
- a singola catena segmentata,
- a doppia catena (dsRNA).
In base alle sue caratteristiche strutturali, il virus ha adottato strategie di replicazione diversificate più o meno lontane dal modello cellulare che vede i passaggi dal DNA all’RNA messaggero (mRNA) e dal RNA messaggero alle proteine con l’intervento dei ribosomi.
In linea di massima i virus a DNA devono penetrare nel nucleo cellulare dove trovano gli enzimi RNA polimerasi DNA dipendente per la sintesi degli RNA messaggeri (dsDNA), i DNA polimerasi che duplicano le catene singole o doppie di DNA virale.
Gli HPV sono virus nudi (senza pericapside), a dsDNA circolare che codifica per sei geni precoci (modificano il metabolismo della cellula infettata e lo mettono al servizio del virus) e due geni tardivi (proteine strutturali). Le proteine codificate dal genoma virale legano e inattivano oncosoppressori come p53 e Rb ed inducono la comparsa di verruche, condilomi e carcinomi della cervice e della vagina.
In rari casi (poxvirus del vaiolo e del mollusco contagioso) nel nucleo virale risiede anche una polimerasi che può attivare tutto il processo sintetico e replicativo nel citoplasma. Alcuni virus (epatite A) hanno il DNA spezzato e richiedono una “riparazione” nel nucleo ad opera di enzimi specifici della cellula ospite.
Molti utilizzano sistemi di replicazione a più tempi (herpes virus, adenovirus), partendo da un mRNA precocissimo producono alcune proteine enzimatiche che consentono di produrre mRNA precoci per altre proteine enzimatiche che leggono mRNA tardivi per la sintesi di proteine tardive strutturali e la replicazione dei filamenti di DNA virali.
Gli ssRNA virus con intermedio a dsDNA sono i retrovirus dell’HIV: nel nucleo virale risiede un enzima detto transcriptasi inversa, in grado cioè di copiare da una catena RNA un filamento di DNA, il quale viene portato nel nucleo, raddoppiato e, tramite le polimerasi cellulari, riletto per la sintesi di mRNA per la proteosintesi e ssRNA per la replicazione del virus.
Gli ssRNA positivi, fungono direttamente da mRNA e possono essere lette dai ribosomi per la sintesi di proteine, fra le quali una RNA polimerasi RNA dipendente, in grado di formare la copia in negativo del filamento virale e da questa le altre copie replicative del virus.
Gli ssRNAvirus negativi (virus dell’influenza) devono essere accompagnati da una RNA polimerasi RNA dipendente funzionante, che sintetizza la copia positiva, un mRNA che viene letto dai ribosomi per la sintesi proteica. Generalmente viene sintetizzata una singola catena polipeptidica che viene scissa nelle proteine funzionali da enzimi citoplasmatici, altrimenti il filamento di RNA può essere segmentato ed ognuno deputato ad una singola proteina.
Per i ribovirus a dsRNA (rotavirus, responsabile delle gastroenteriti e, spesso, del diabete mellito tipo 1 dei bambini). Nel nucleo virale è presente una RNA polimerasi RNA dipendente per iniziare il ciclo replicativo. Solo la catena (-)RNA del genoma viene trascritta per formare molecole di (+)RNA che hanno la duplice funzione di mRNA e di “stampo” per la sintesi di molecole di (-)RNA complementari: (+)RNA e (-)RNA rimangono uniti generando il dsRNA del genoma virale
Il coronavirus rilascia il suo (+)ssRNA all’interno del citoplasma della cellula che viene tradotto dai ribosomi, con la produzione di una RNA-polimerasi RNA-dipendente (proteina L) che trascrive un (-)ssRNA, che funge da “stampo” da cui poi è possibile ottenere nuovi (+)ssRNA del coronavirus, nonché le sette proteine che esso codifica. A ciascun nuovo filamento di RNA positivo viene associata come stabilizzatrice la proteina N, mentre le proteine del pericapside si fondono alla membrana del reticolo endoplasmatico e passano nel lume. Successivamente dal reticolo endoplamatico gemmano i nuovi virioni per essere rilasciati mediante esocitosi, portandosi via il rivestimento di membrana cellulare ospite.
La struttura del DNA e dell’RNA
Il DNA e l’RNA sono acidi nucleici formati da catene di nucleotidi, ognuna delle quali è costituito da un gruppo fosfato, uno zucchero a 5 atomi di carbonio ed una base azotata.
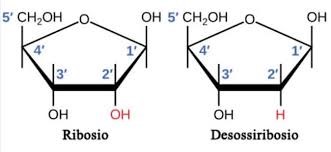
Nel DNA lo zucchero è il desossiribosio, che può essere legato a quattro basi azotate differenti: adenina (A); timina (T); guanina (G); citosina (C). Nell’RNA lo zucchero è il ribosio mentre delle basi la timina è sostituita dall’uracile (U).
L’adenina e la guanina derivano dalla purina, la citosina, la timina e l’uracile derivano dalla pirimidina, entrambi composti eterociclici.
Il gruppo fosfato di ogni nucleotide lega il carbonio 3’ di uno zucchero al 5’ del nucleotide seguente per mezzo di uno stabile legame covalente.
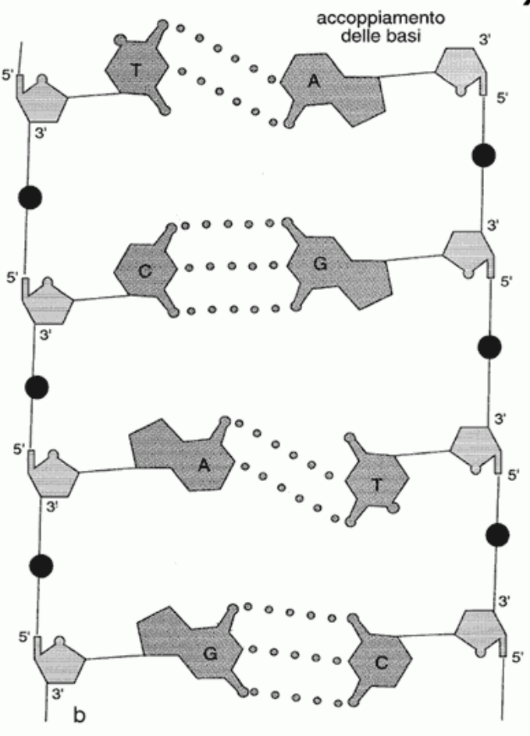
Il DNA a doppia catena si unisce ad una catena complementare dove il gruppo fosfato lega il carbonio 5’ al 3’ di un altro nucleotide, con un orientamento opposto mentre l’adenina forma due legami deboli con la timina e la guanina tre legami con la citosina.
Il codone di inizio è generalmente AUG = metionina, mentre i codoni finali di blocco, UAG, UAA e UGA, sono non-sense perché non rappresentano nessun aminoacido ed interrompono la sintesi proteica, tutte le triplette del DNA rappresentano codici identificativi di aminoacidi. Con 64 possibili combinazioni, ciascuno dei 21 aminoacidi può essere codificato da più di una tripletta.
Della doppia elica che costituisce il DNA, una viene letta e trascritta in senso 5’-3’ mediante la RNA polimerasi nei mRNA, mentre l’altra può essere letta per altri geni; la doppia elica serve a stabilizzare il DNA e mantenere uno stampo per la riparazione in caso di errori o perdite di nucleotidi.
Esistono tre tipi diversi di RNA:
- RNA messaggero, o mRNA, che riproduce l’esatta sequenza di aminoacidi di ogni proteina da portare nel citoplasma;
- RNA di trasporto, o tRNA, ha una forma a trifoglio che aggancia un aminoacido e presenta la tripletta complementare (anticodone) che identifica l’aminoacido nel mRNA;
- RNA ribosomiale, o rRNA, sintetizzato nel nucleolo da una delle parti più stabili dei cromosomi. Quantitativamente rappresenta l’80% di tutto l’RNA citoplasmatico.
Il mRNA viene agganciato dal rRNA di un ribosoma che comincia a scorrerlo “leggendo” le triplette e consentendo l’interazione con l’anticodone dell’RNA transfer portatore dell’aminoacido corrispondente che viene legato ai successivi fino a formare la catena proteica (vengono montati due aminoacidi al secondo).
La struttura “desossi” rende stabile la molecola di DNA, mentre il ribosio, con il terminale OH sul carbonio 2’ tende a reagire con vari elementi chimici e risulta quindi più instabile. Il fatto poi che la catena RNA sia solitamente singola la espone a frequenti mutazioni che avvengono in corso di trascrizione degli RNA virali, con modifica delle caratteristiche antigeniche dei capsomeri e dei peplomeri. Questo determina la perdita dell’immunità da precedenti infezioni dello stesso virus e la sua possibilità di penetrare in cellule diverse (mucose respiratorie, fegato, cellule pancreatiche, encefalo, ecc.) o di specie diverse (spillover).
I farmaci antivirali noti agiscono bloccando gli enzimi virali, purtroppo il farmaco attivo sulla polimerasi influenzale non è efficace contro altre polimerasi: ogni enzima può avere un suo antivirale.
Esistono diversi tipi di farmaci antivirali, ognuno dei quali specifico per combattere un particolare tipo di virus:
- Farmaci antivirus influenzali: amantadina, in grado di contrastare il virus influenzale A; zanamivir e oseltamivir attivi contro i virus influenzali di tipo A e B, compresi i ceppi resistenti all’amantadina;
- Farmaci antivirus erpetici: aciclovir è l’antivirale di riferimento per la terapia anti-herpes. Trova impiego sia contro l’Herpes labiale e genitale (Herpes simplex) che contro la varicella e l’Herpes-zoster. Ci sono poi valaciclovir, penciclovir e famciclovir che hanno uno spettro d’azione simile all’aciclovir, ma maggiori effetti collaterali. Contro questi virus possono essere utilizzati anche l’idoxuridina e la vidarabina (quest’ultima soprattutto contro l’Herpes simplex);
- Farmaci antivirus epatici: interferoni, per il trattamento di diversi tipi di epatiti e la ribavirina, utilizzata nell’epatite cronica da virus dell’epatite C;
- Farmaci anti HIV: la terapia HAART, Highly Active Anti-Retroviral Therapy, ovvero terapia antiretrovirale altamente attiva consiste nell’uso associato di più antivirali nella terapia dell’infezione da HIV: questa associazione è necessaria poiché con la monoterapia questo virus diventa facilmente resistente. Tra gli antivirali utilizzati contro il virus dell’HIV ci sono la zidovudina o AZT, il fosamprenavir e il metilsoprinolo.
- Farmaci antivirus della famiglia Poxviridae: trifluridina e metisazone (le infezioni causate dai poxvirus determinano generalmente la comparsa di lesioni cutanee nodulari o pustolose).
Spesso il farmaco rallenta la replicazione e dovrà essere l’organismo, con gli anticorpi che va producendo, a debellare completamente l’infezione. In molti casi il virus rimane intracellulare e si riattiva ogni volta che le difese immunitarie si abbassano per altre patologie intercorrenti.
Indubbiamente il trattamento più efficace è l’individuazione di un vaccino che consenta all’organismo di bloccare il virus subito in fase iniziale, prima che abbia determinato gravi danni ai vari organi.
Risposte immunitarie innate
l macrofagi inglobano i virus e altri agenti patogeni che sono stati neutralizzati dagli anticorpi circolanti e che devono essere distrutti senza danneggiare i tessuti circostanti. l macrofagi derivano dai monociti del sangue che si differenziano in macrofagi nei polmoni, nei linfonodi, nella milza, nella cute, nel sistema nervoso centrale (dove formano la microglia) e nel fegato.
Dopo aver inglobato la particella, il macrofago digerisce il contenuto della vescicola endocitata. l prodotti di digestione della particella vengono trasportati alla superficie cellulare in associazione con il complesso maggiore di istocompatibilità (MHC, Major Histocompatibility Complex) in grado di attivare i linfociti T helper
I macrofagi sono efficienti non solo nel digerire antigeni e nel presentare componenti estranee, ma anche nel produrre alcune citochine, fra cui le interleuchine 1 e 6. Con queste un macrofago infetto allerta perciò le cellule circostanti, inclusi i linfociti B, della presenza di materiale estraneo potenzialmente infettivo.
L’interleuchina 6 è anche una “miochina”, ovvero una citochina prodotta dal muscolo, e la sua concentrazione aumenta in risposta alla contrazione e precede la comparsa in circolo di altre citochine. Verosimilmente durante l’esercizio fisico determina una vasodilatazione che favorisce l’apporto di sostanze nutritive al muscolo.
La IL-6 è anche uno dei più importanti mediatori della febbre in quanto è in grado di attraversare la barriera emato-encefalica e agire sull’ipotalamo.
Un eccesso di IL-6 può determinare una congestione locale e compromissione dell’organo in cui questo avviene.
L’interferone α/β è prodotto da una grande varietà di cellule in seguito all’infezione virale e attiva la citotossicità mediata dalle cellule NK tramite il legame a specifici recettori. Alcuni virus sono in grado di produrre una molecola solubile analoga al recettore dell’interferone di tipo I che inibisce l’effetto antivirale dell’interferone stesso, competendo con i normali meccanismi di attivazione.
Le cellule NK sono reclutate entro i primi due giorni dall’infezione e si dividono e si differenziano localmente. Esse hanno importanti effetti antivirali, in parte perché uccidono le cellule infettate attraverso la formazione di granuli citolitici e in parte perché producono interferoni α, β e γ.
Risposte immunitarie acquisite
I linfociti specifici per l’antigene proliferano e si differenziano durante questo intervallo di tempo, raggiungendo valori che sono sufficienti a costituire una difesa efficace mediante la produzione di anticorpi sempre più perfezionati. Se si è incontrato un agente patogeno simile in precedenza o si è stati vaccinati contro tale patogeno, il ritardo si riduce e la potenza della risposta immunitaria è fortemente rafforzata; questo fenomeno viene definito memoria immunitaria.
Per approfondimenti:
R. Burioni: “VIRUS la grande sfida. Dal coronavirus alla peste: come la scienza può salvare l’umanità”. Rizzoli, marzo 2020.
Medical Facts: www.medicalfacts.it
